Manipulação de dados tabulares - Pandas e alternativas
Pandas, SQL e comparações (Dask, Polars, Datatable, PandaSQL e DuckDB)
Introdução
Em machine learning, a gente sempre comenta como nós precisamos de dados. E precisamos de dados bem curados. Talvez você até já tenha ouvido a frase: "garbage in, garbage out", né?
Na vida real, isso é bem verdade. E gastar mais tempo criando novas features, ou limpando os dados acaba muitas vezes impactando os resultados dos modelos mais que qualquer outra coisa. Não é à toa que grande parte dos ganhadores das competições do Kaggle usam os mesmo modelos - ensembles. E, na maior parte, quem ganha são os que tem os melhores dados.
Então, antes de mexermos em modelos, temos que manipular os dados. Mas então como manipular esses dados?
Pandas
A resposta mais comum é pandas! Se você já mexeu com dados, provavelmente já ouviu falar sobre essa biblioteca. Ela é, de longe, a mais popular pra manipulação de dados tabulares. Ela foi construída em cima de NumPy, que usa C++ por trás dos panos.
Em Python temos o GIL que limita o uso de threads pra evitar que mais de um thread tente escrever/apagar as mesmas partes da memória ao mesmo tempo (condição de corrida). Quando usamos Numpy e chegamos no nível de C, o programa não está sujeito ao GIL e então podemos parallelizar as coisas. Mas se você já ouviu falar de pandas, é capaz que não tenha ouvido coisas boas. Especialmente quem vem da linguagem de programação em R usando o dplyr tem o que falar 😅. O número de perguntas no StackOverflow relacionadas à pandas cresceu bastante durante os anos. Parte pelo aumento em popularidade da biblioteca, mas parte pela dificuldade da API do pandas.
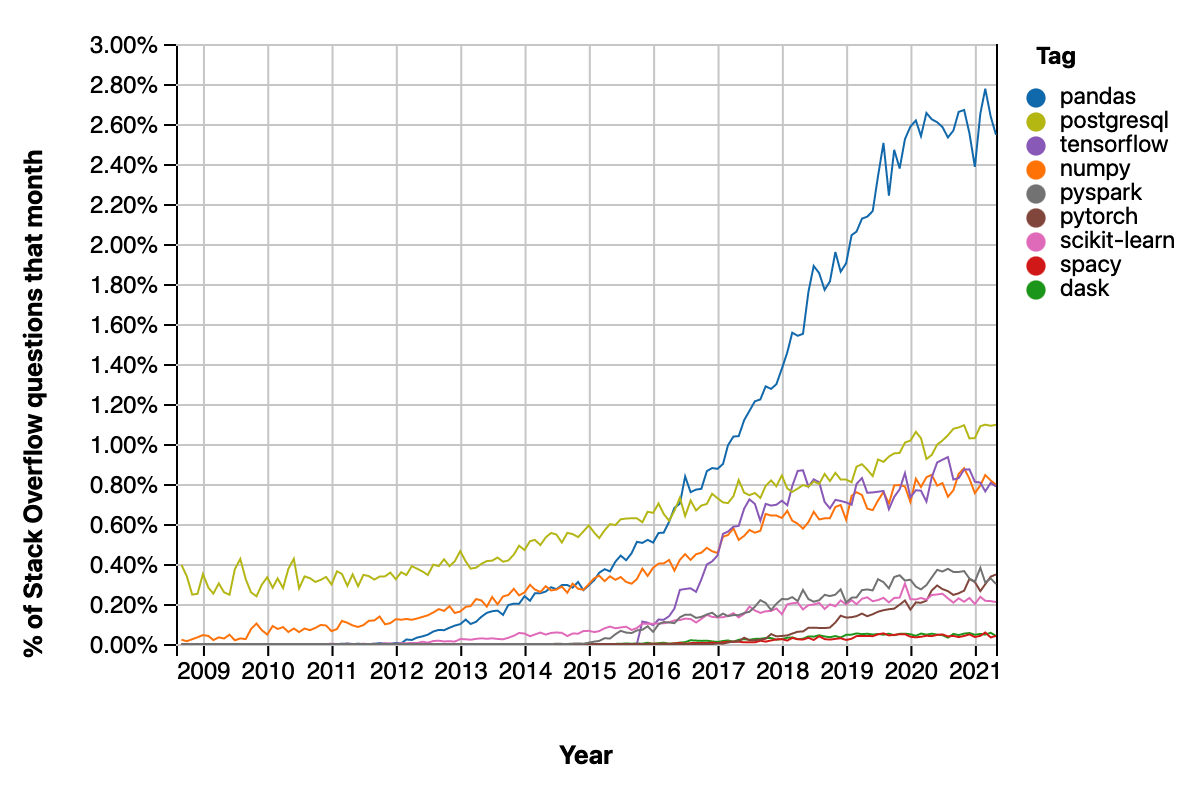
Além disso, existem vários pontos em que podemos otimizar a performance de pandas. Isso pelo fato da biblioteca ter sido criada em cima de uma outra. Vamos dar uma olhada na performance de pandas.
Dados
Vamos usar o TPC-H dataset, focando no lineitem e orders, que são as maiores tabelas que somam 1GB. Para medir o tempo de execução, vamos unir as duas tabelas, filtram as linhas, agrupam os dados e computamos alguns dados agregados (máximo, mínimo, etc.). O que estamos fazendo em si não é muito importante, mas é um tipo de transformação que encontraríamos na vida real.
Vamos tentar otimizar tudo ao máximo, e vamos focar na transformação dos dados
- Vamos medir o tempo da transformação (excluindo o tempo de ler os dados)
- Vamos filtrar o quanto antes
- Vamos encadear operações
%%time
lineitem.merge(orders, left_on="l_orderkey", right_on="o_orderkey").pipe(
lambda df: df.copy()[
(df["l_shipdate"] < "1998-09-02")
& (df["o_orderpriority"].isin(("1-URGENT", "2-HIGH")))
]
).groupby(["l_returnflag", "l_linestatus"]).agg(
{
"l_extendedprice": ["sum", "min", "max", "mean"],
"l_quantity": ["sum", "min", "max", "mean"],
}
)
Executando todas essas transformações demorou 10.3 segundos. Mas podemos ser mais eficientes. Por exemplo, poderíamos esquecer várias colunas já que no fim só estamos interessados em l_returnflag, l_linestatus, l_extendedprice e l_quantity (além das colunas que usamos para filtrar os dados). Também somos ineficientes quando temos de filtrar e agregar os valores (escaneamos a tabela duas vezes, quando poderíamos fazer isso de uma vez só). Além disso, poderíamos melhorar na implementação de paralelismo e do algoritmo.
Vamos então deixar pandas de lado e ver como podemos melhorar isso.

Dask
Dask provavelmente é a primeira solução que vem à cabeça. Dask basicamente cria várias tabelas em pandas e vai paralelizando as operações em cores diferentes. Também é mais eficiente por usar avaliação preguiçosa (lazy evaluation) - primeiro traçamos o que queremos fazer e depois mandamos o computador executar as transformações. Isso nos deixa otimizar a computação das transformações (por exemplo, podemos filtrar linhas antes de fazer qualquer outra coisa, mesmo que o código não represente isso).

# collapse
# dask
dd_lineitem = dd.from_pandas(lineitem, npartitions=1)
dd_orders = dd.from_pandas(orders, npartitions=1)
%%timeit
dd_lineitem.merge(dd_orders, left_on="l_orderkey", right_on="o_orderkey").pipe(
lambda df: df.copy()[
(df["l_shipdate"] < "1998-09-02")
& (df["o_orderpriority"].isin(("1-URGENT", "2-HIGH")))
]
).groupby(["l_returnflag", "l_linestatus"]).agg(
{
"l_extendedprice": ["sum", "min", "max", "mean"],
"l_quantity": ["sum", "min", "max", "mean"],
}
).compute()
Usando Dask e com essas melhorias de parallelismo a gente ganha alguns segundos. O que faz Dask muito popular é o fato de ele imitar a API do Pandas. Aí fica muito mais fácil de pegar e começar a codar. Mas ainda assim, estamos usando Pandas. Temos espaços para melhorias.
Polars
Polars é uma biblioteca implementada em Rust, que é muito eficiente em termos de velocidade e uso de memória. O lugar de Polars (de acordo com a documentação) é para quando os dados são muito grandes para Pandas mas muito pequenos para realizar as transformações num cluster de máquinas (Spark). Ou seja, se os dados são muito grandes pra sua máquina, mesmo depois de filtrar algumas entradas, precisamos procurar outras soluções (o que é verdade pra Dask e todas as outras bibliotecas que vamos ver aqui). E assim como Dask, Polars vai usar todas as threads da sua máquina.
Uma outra idéia de Polars é permitir o use da avaliação ansiosa (eager evaluation), como em Pandas e da avaliação preguiçosa (lazy evaluation), como em Dask. A availação ansiosa é como em Pandas, e a API é bem parecida. A API da avaliação preguiçosa é um pouco diferente, e é otimizada pelos mesmo motivos que citamos quando falamos de Dask (paralelização e otimização das transformações).

# collapse
# polars
pl_lineitem = pl.DataFrame(lineitem)
pl_orders = pl.DataFrame(orders)
%%timeit
pl_lineitem.join(pl_orders, left_on="l_orderkey", right_on="o_orderkey").filter(
(pl.col("l_shipdate") < "1998-09-02")
& (
(pl.col("o_orderpriority") == "1-URGENT")
| (pl.col("o_orderpriority") == "2-HIGH")
)
).groupby(["l_returnflag", "l_linestatus"]).agg(
{
"l_extendedprice": ["sum", "min", "max", "mean"],
"l_quantity": ["sum", "min", "max", "mean"],
}
)
Com isso conseguimos reduzir o tempo em <30% (comparando com Dask)! Então, o que ainda podemos melhorar? Quando usamos a avaliação ansiosa, ainda podemos melhorar a execução das queries. Além disso, a biblioteca não tem tantas funções quanto Pandas (e consequentemente Dask). E falando em Pandas, se a API é igual, e você não está satisfeito com ela, provavelmente não vai estar muito contente com a API do Polars também.
Datatable
Se você gosta de R e está fazendo a transição para python, talvez essa seja uma boa opção. Datatable é inspirada pela biblioteca em R data.table. A API é parecida, e tem várias otimizações para tratar com dados grandes. Que cabem ou não em memória (dados que não caberiam em memória usando Pandas cabem quando usamos Datatable). Essa biblioteca também usa algoritmos que supportam o paralelismo em diferentes threads), aumentando de novo a velocidade de execução.

# collapse
# datatable
dt_lineitem = dt.Frame(lineitem)
dt_orders = dt.Frame(orders)
# preparação
dt_lineitem.names = {"l_orderkey": "orderkey"}
dt_orders.names = {"o_orderkey": "orderkey"}
%%timeit
# dt_lineitem.key = "orderkey" # não podemos usar como `key` existem valores repetidos
dt_orders.key = "orderkey"
# executando a query
dt_lineitem[
(g.o_orderpriority == "1-URGENT") | (g.o_orderpriority == "2-HIGH"),
:,
join(dt_orders),
][
:,
{
"sum(l_extendedprice)": dt.sum(f.l_extendedprice),
"min(l_extendedprice)": dt.min(f.l_extendedprice),
"max(l_extendedprice)": dt.max(f.l_extendedprice),
"mean(l_extendedprice)": dt.mean(f.l_extendedprice),
"sum(l_quantity)": dt.sum(f.l_quantity),
"min(l_quantity)": dt.min(f.l_quantity),
"max(l_quantity)": dt.max(f.l_quantity),
"mean(l_quantity)": dt.mean(f.l_quantity),
},
by(f.l_returnflag, f.l_linestatus),
]
~10% do tempo (comparando com Pandas)! Você pode reparar também como a API muda bastante. Pessoalmente, foi um pouco mais trabalhoso pra escrever o código equivalente, mas na documentação eles também ofereçem uma comparação da API do Datatable com a API do Pandas, o que ajuda bastante.
Além disso, eles também comparam a API do Datatable com SQL. Afinal de contas, SQL é muito usado até hoje, e mais familiar pra muitas pessoas. Além disso, SQL tem sido usado bastante através dos anos e oferece uma maneira eficiente de manipular dados tabulars. Quando os dados aumentam ainda mais e nos tornamos para um método distribuído como Spark ainda podemos usar SparkSQL. Spark e SQL precisam de um cluster e de um servidor) próprio pra executar as transformações. Mas existem algumas bibliotecas que nos permitem usar SQL para manipular dados em dataframes.
PandaSQL
PandasSQL é uma solução mais antiga. A biblioteca permite usar SQL (SQLite) em tabelas Pandas. Você só tem que passar a query em SQL e as variáveis locais. Por trás dos panos, eles criam um SQL engine usando SQLAlchemy e os métodos de to_sql e read_sql de Pandas. O que faz possível executar queries em Pandas, mas é um truque, que acaba sendo bem ineficiente em relação a tempo de execução e uso de memória #gambiarra

# hide_output
from pandasql import sqldf
start = time.monotonic()
_df = sqldf(
"""
SELECT l_returnflag,
l_linestatus,
SUM(l_extendedprice),
MIN(l_extendedprice),
MAX(l_extendedprice),
AVG(l_extendedprice),
SUM(l_quantity),
MIN(l_quantity),
MAX(l_quantity),
AVG(l_quantity)
FROM lineitem AS lineitem
JOIN orders AS orders ON (l_orderkey=o_orderkey)
WHERE l_shipdate <= DATE '1998-09-02'
AND o_orderpriority IN ('1-URGENT', '2-HIGH')
GROUP BY l_returnflag,
l_linestatus
""",
globals(),
)
# o código retorna um erro interno (`OperationalError`) - visite o notebook para mais detalhes
print(f"Wall time: {time.monotonic() - start:.2f}s")
_df
Não muito bom... Mas deve existir outro jeito de usar SQL em Pandas, e ainda aproveitando a eficiência de SQL, não? Na verdade sim. Acontece que tem gente que basicamente reconstruiu um servidor de SQL pra ser executado em memória, com uma API em python!
DuckDB
DuckDB permite aos desenvolvedores à executar queries nas suas tabelas em Pandas ou direto de arquivos CSV, parquet, etc. A biblioteca foi desenvolvida com C++ por trás dos panos, e também permite paralelização e otimização da query. Ou seja, eles evitam problemas que encontramos em outras bibliotecas - antes nós tinhamos de escanear a tabela duas vezes quando executamos nossa query: uma para filtrar as linhas e outra vez pra agroupar os dados. Isso não acontece com DuckDB. Eles também são inteligente no uso de memória.
Também pensando no ambiente python, onde Pandas ainda é prevalente, eles oferecem métodos para transformar os dados numa tabela Pandas.

# ler arquivos em parquet e colocar em uma dataframe
lineitem = duckdb.query("SELECT * FROM 'lineitemsf1.snappy.parquet'").to_df()
orders = duckdb.query("SELECT * FROM 'orders.parquet'").to_df()
lineitem.head()
%%timeit
# executar uma query na nossa tabela Pandas
duckdb.query(
"""
SELECT l_returnflag,
l_linestatus,
SUM(l_extendedprice),
MIN(l_extendedprice),
MAX(l_extendedprice),
AVG(l_extendedprice),
SUM(l_quantity),
MIN(l_quantity),
MAX(l_quantity),
AVG(l_quantity)
FROM lineitem AS lineitem
JOIN orders AS orders ON (l_orderkey=o_orderkey)
WHERE l_shipdate <= DATE '1998-09-02'
AND o_orderpriority IN ('1-URGENT', '2-HIGH')
GROUP BY l_returnflag,
l_linestatus
"""
).to_df()
Além de ser a mais rápida, repare que DuckDB já pega as variáveis locais quando tentamos executar as queries. A biblioteca também suporta (código testado) vários tipos de SQL, incluindo PostgreSQL, MySQL e SQLite.
Mais informações
O DuckDB foi a solução mais rápida. Mas como você deve ter imaginado, esse não é sempre o caso. Tudo depende do volume de dados que temos e das transformações que vamos fazer. Os criadores do Datatable também comparam o tempo de execução das queries para diferentes cenários. As comparações são atualizadas com o passar do tempo e você pode encontrar os últimos resultados aqui.
Para os interessados, também tem um artigo muito interessante no blog do DuckDB, onde eles explicam um pouco os motivos quais DuckDB tem uma performance elevada à Pandas, e como que podemos otimizar nossas queries (mesmo que só em Pandas). Esse post também foi inspirado no blog post deles (as queries e os dados). Vale à pena dar uma olhada.
Quando falamos de Big Data também não podemos deixar de falar de outras tecnologias, como Spark, Apache Beam, etc. Quando os dados são muito grandes pra uma máquina só, o que nos resta é utilizar um cluster de computadores e paralelizar as computações e dividir o uso de memória - escalabilidade horizontal. Mas com esses clusters precisamos de uma parte administrativa maior, e não seria algo que qualquer um poderia testar rapidamente. Nesse post incluímos apenas os casos de quando os dados caberiam em uma máquina, e estamos avaliando a eficiência de bibliotecas em python (existem outras bibliotecas em outras linguagens que também são bem eficientes).
Em outras palavras, nós comparamos algumas ferramentas pra um específico caso, mas pra determinar a melhor ferramenta, precisamos ir de caso-a-caso. Qual o volume de dados? Precisamos um cluster? Qual o tempo de execução aceitável? Qual o nível técnico da equipe?